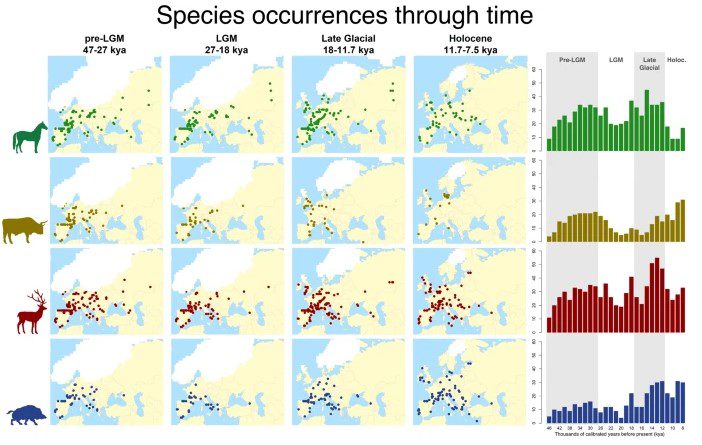
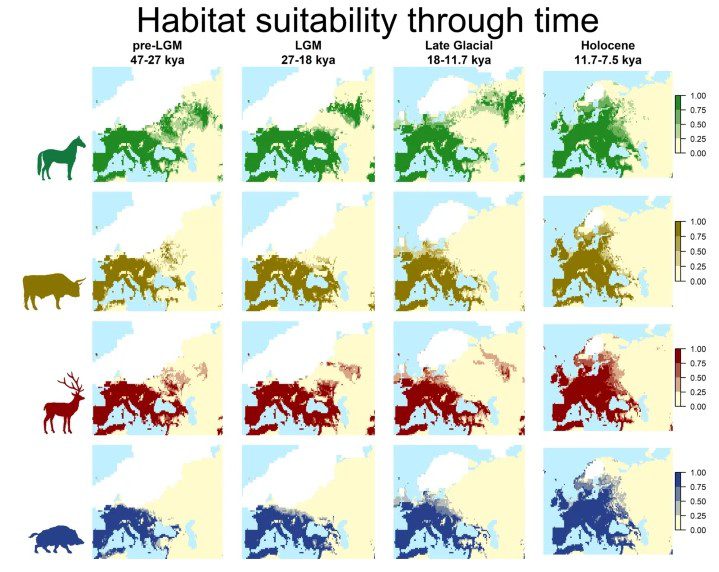
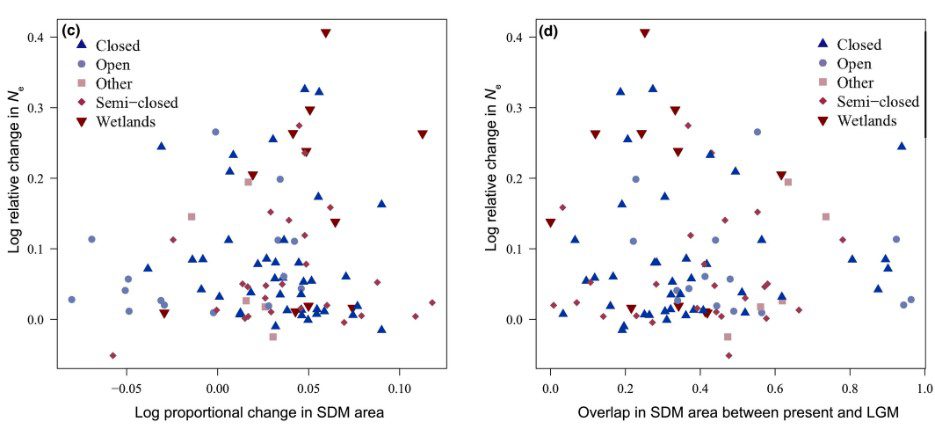
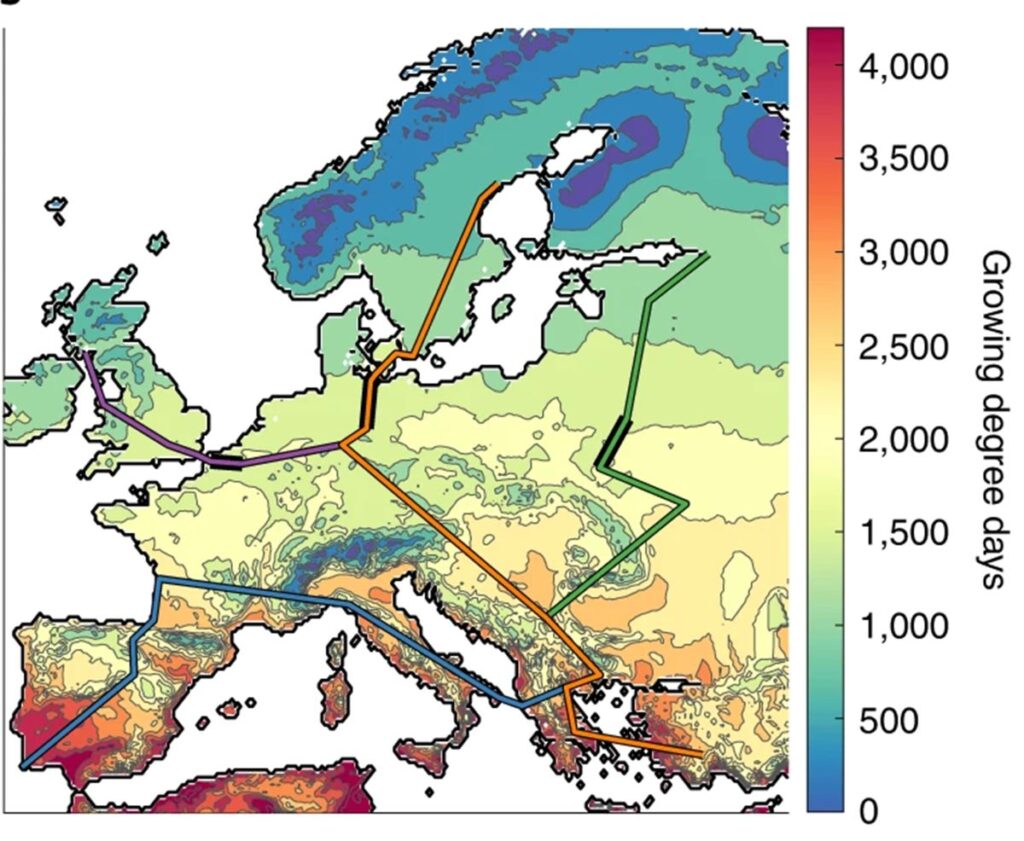
pastclim, il nostro pacchetto R per facilitare l’uso e la manipolazione di dati paleoclimatici è stato aggiornato alla versione 1.2. Inoltre, è uscito su Ecography l’articolo che lo descrive:
Michela Leonardi, Emily Y. Hallett, Robert Beyer, Mario Krapp, Andrea Manica
pastclim 1.2: an R package to easily access and use paleoclimatic reconstructions
Ecography, First published: 05 January 2023 https://doi.org/10.1111/ecog.06481

Ecco le novità rispetto alla versione presentata nel preprint:
- pastclim è ora disponibile su CRAN: https://cran.r-project.org/web/packages/pastclim/index.html;
- Abbiamo aggiunto funzioni per ritagliare i dati sulla base di rettangoli o maschere, e per campionare punti randomicamente nello spazio e nel tempo;
- Abbiamo creato un cheatsheet, un’infografica che riassume le funzioni principali;
- Un nuovo set di istruzioni spiega come lavorare con dati paleoclimatici non inclusi nel pacchetto, e come aggiungerli al pacchetto per renderli disponibili a tutti gli utenti.
ed ecco una tabella riassuntiva delle funzioni presenti nel pacchetto
| Download the data | |
| get_data_path() | Retrieve the path in which pastclim automatically stores the data |
| set_data_path() | Store the data in a custom path |
| download_dataset() | Download a whole dataset (all variables available) |
| get_vars_for_dataset() Download variables of choice for a given dataset | Download variables of choice for a given dataset |
| get_downloaded_datasets() | Summary of the downloaded variables |
| get_time_steps() | List of time steps available in a given dataset |
| Get climate for locations or regions | |
| location_slice() | Get the climate for given locations (by coordinates and age) |
| location_series() | Get time series of the climate for given locations |
| region_slice() | get the climate for a given region in a given time step |
| sample_region_slice() | sample a given number of points from the climate of a region |
| region_series() | get the time series of the climate for a given region |
| sample_region_series() | sample a given series of points from the time series of a region |
| Working with biomes and ice sheets | |
| get_biome_classes() | legend of the ‘biome’ categorical variable, when available |
| get_ice_mask() | get a mask with the extent of the ice sheets for a given time step |
| get_land_mask() | get a mask with the extent of the land masses for a given time step |
| Vignettes | |
| vignette(“pastclim_overview”, package = “pastclim”) | overview of pastclim |
| vignette(“custom_datasets”, package = “pastclim”) | how to add a new dataset to pastclim |
| vignette(“available_datasets”, package = “pastclim”) | list of datasets available |
Per maggiori informazioni:
sito web: https://evolecolgroup.github.io/pastclim/index.html
istruzioni: https://rdrr.io/github/EvolEcolGroup/pastclim/
repository github: https://github.com/EvolEcolGroup/pastclim
CRAN: https://cran.r-project.org/web/packages/pastclim/index.html
vignette: https://evolecolgroup.github.io/pastclim/articles/a0_pastclim_overview.html
cheatsheet: evolecolgroup.github.io/pastclim/pastclim_cheatsheet.pdf